2. 様々なデータベースと配列解析
2-2.遺伝子領域、機能の予測 その1
■ 相同性検索
生物は少しずつ DNA を変化させて進化してきました。そのため近縁種の生物であるほど、
そのゲノム配列は高い類似性を持っています。 このことから、ある2種類の生物の遺伝子を比較してその配列が似ているならば、
両者は進化的に近い関係があると考えられます。
さらに、遺伝子やコードされているタンパク質の機能、構造も似ていると考えられます。
この考えに基づき、機能が未知である配列などに対して高い類似性を持つ配列をデータベースから検索し、 機能や立体構造を推測するという手法が用いられています。この検索を一般に相同性検索(ホモロジー検索)といいます。
この考えに基づき、機能が未知である配列などに対して高い類似性を持つ配列をデータベースから検索し、 機能や立体構造を推測するという手法が用いられています。この検索を一般に相同性検索(ホモロジー検索)といいます。
■ 相同性検索プログラム
「類似性の高い配列をデータベースから検索する。」と言うのは簡単ですが、
「似ているもの」を探すようにコンピュータにお願いすることはそれほど単純ではありません。
コンピュータでは、基本的に if 文などによって「同じかどうか」という比較しかできないからです。
<似ている配列>
例1)配列の一部が異なる
配列1 AAAACGTGGGGG (データベース内の配列)
配列2 ACCTG (調べたい配列:質問配列、クエリ)
例2)欠損または挿入がある(ギャップ)
配列1 AAAAC-TGGGGG
配列2 ACCTG
このような相同性検索のための代表的なプログラムとして、以下のものがあります。
コンピュータでは、基本的に if 文などによって「同じかどうか」という比較しかできないからです。
<似ている配列>
例1)配列の一部が異なる
配列1 AAAACGTGGGGG (データベース内の配列)
配列2 ACCTG (調べたい配列:質問配列、クエリ)
例2)欠損または挿入がある(ギャップ)
配列1 AAAAC-TGGGGG
配列2 ACCTG
このような相同性検索のための代表的なプログラムとして、以下のものがあります。
- FASTA(Pearson 1988:米 Virginia Univ.): 検出感度は BLAST より優れている。
- NCBI-BLAST(Altschul 1990:米 NCBI): FASTA より高速。現在、もっともよく使われている。
- PSI-BLAST(Altschul 1997): BLAST の結果(複数の配列)をクエリとして検索し、その結果を用いてさらに検索する。 結果が変わらなくなるまで繰り返す。
■ BLAST の実行
BLAST は、世界中でもっともよく利用されている相同性検索プログラムであり、
NCBI、
GenomeNet、
DDBJ 等のウェブサイトで利用することができます。
検索する際には、指定しなければいけない項目がいくつかあります。
検索する際には、指定しなければいけない項目がいくつかあります。
-
Query Sequence(アミノ酸配列または塩基配列)
BLAST に限らず多くの配列解析プログラムに渡す配列は、FASTA 形式にしておく必要があります。 FASTA 形式は、一行目に ">" で始まるコメントがあり、2行目以降に配列が来ます。
> BAA81644.1
MLDQKTIDIIKSTVPVLKSNGLEITKTFYKNMFEQNPEVKPLFNMNKQESEEQPKALAMA
ILAVAQNI・・・・・・・
・・・・・・
-
Program
相同性検索をする際のクエリ配列と対象データベースの組み合わせによってプログラムを選択します。
クエリおよびデータベースがアミノ酸配列の場合は、blastp を使用します。検索プログラム クエリ配列 対象データベース 比較する配列レベル blastn 塩基配列 塩基配列 塩基配列同士を比較 blastx 塩基配列 アミノ酸配列 翻訳したクエリ配列を使用して検索を行う blastp アミノ酸配列 アミノ酸配列 アミノ酸配列同士を比較 tblastn アミノ酸配列 塩基配列 データベースを翻訳しながら検索を行う tblastx 塩基配列 塩基配列 翻訳したアミノ酸配列同士を比較 -
Database
選択できるデータベースは、検索サイトによって多少ことなる部分もありますが、 nr や nt、 PDB などはどのサイトでも利用できます。データベース 内容 nr 複数のデータベースを統合し、重複を除いたアミノ酸配列データベース nt 複数のデータベースを統合し、重複を除いた塩基配列データベース PDB 立体構造既知のタンパク質のアミノ酸配列データベース KEGG GENES Genome
■ BLAST の結果
Score 値が大きく、E-value (期待値)が小さいと、有意な相同性があると判定され、リストの上の方へ来ます。
スコアは、配列要素ごとに定義される類似度のスコアの和として計算されます。
E-value は、相同性のある配列が偶然に見つかる場合の期待値のことです。この値が低いほど、
その相同性が必然的であることを示しています。
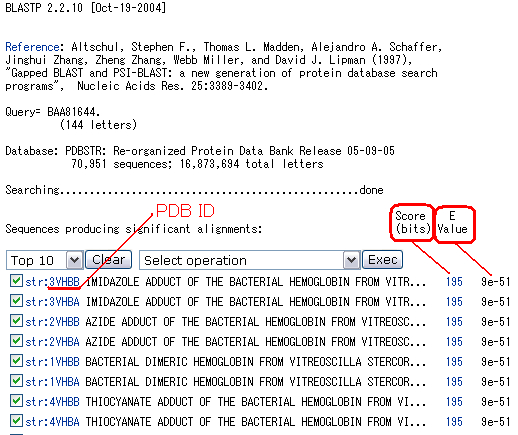

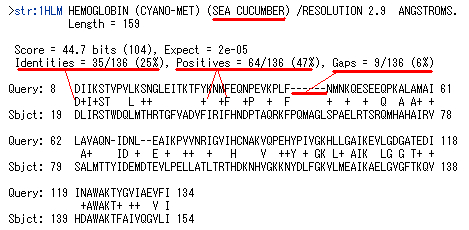
リストの下には、クエリ配列とデータベース内の配列とを比較したアライメントがリストの順に並んでいます。
スコア値をクリックすると対応するアライメントにジャンプすることができます。図は、スコア値が 45 の配列です。
Identities は、完全に一致した配列要素の割合、Positives は、完全に一致したものに加えて、
プラス評価されている配列要素も含めた割合です。

[ 演習 2-2 ]
演習 1-1 で調べた遺伝子 AB028630 の 2 つ目のタンパク質と配列に類似性のある立体構造既知のタンパク質を検索しよう。 検索サイトは、GenomeNet を利用しよう。 (ヒント!)
演習 1-1 で調べた遺伝子 AB028630 の 2 つ目のタンパク質と配列に類似性のある立体構造既知のタンパク質を検索しよう。 検索サイトは、GenomeNet を利用しよう。 (ヒント!)
■ マルチプルアライメント、系統樹(ClustalW)
GenomeNet のサイトでは、BLAST の結果からいくつかを選んで(チェックボックスにチェックする)、
複数の配列を同時にアライメントするマルチプルアライメントを行うことができます。
もっともよく利用される ClustalW を紹介します。
操作は簡単です。既に得られている BLAST の結果の上の方にある "Select operation" プルダウンを図のように "CLUSTALW"とし、 実行ボタン "Exec" をクリックするだけです。

選択する配列を20配列くらい飛ばしたほうが、マルチプルアライメントしている様子がわかりやすいかもしれません。

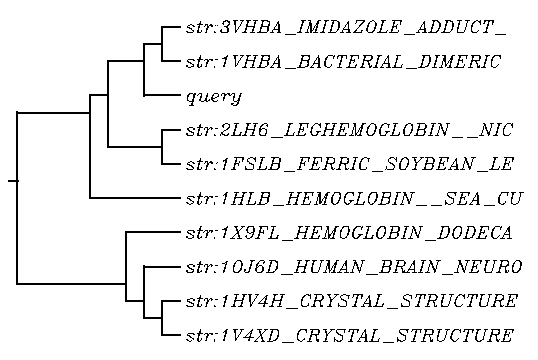
さらに、マルチプルアライメントの結果の一番下にある "Select tree menu" から "N-J Tree" (Neighbor-Joining method : 近隣結合法)を選択し、 実行ボタン "Exec" をクリックすると、 系統樹が作製されます。
もどる
操作は簡単です。既に得られている BLAST の結果の上の方にある "Select operation" プルダウンを図のように "CLUSTALW"とし、 実行ボタン "Exec" をクリックするだけです。

選択する配列を20配列くらい飛ばしたほうが、マルチプルアライメントしている様子がわかりやすいかもしれません。

さらに、マルチプルアライメントの結果の一番下にある "Select tree menu" から "N-J Tree" (Neighbor-Joining method : 近隣結合法)を選択し、 実行ボタン "Exec" をクリックすると、 系統樹が作製されます。


3VHB: Imidazole Adduct Of The Bacterial Hemoglobin From Vitreoscilla Sp.
1VHB: Vitreoscilla Stercoraria
query
2LH6: Lupinus luteus キバナノハウチワマメ
1FSL: soybean 大豆
1HLB: sea cucumber ナマコ
1X9F: Lumbricus terrestris ミミズ
1OJ6: Human
1HV4: Bar-Head Goose ガチョウ
1V4X: bluefin tuna クロマグロ
1VHB: Vitreoscilla Stercoraria
query
2LH6: Lupinus luteus キバナノハウチワマメ
1FSL: soybean 大豆
1HLB: sea cucumber ナマコ
1X9F: Lumbricus terrestris ミミズ
1OJ6: Human
1HV4: Bar-Head Goose ガチョウ
1V4X: bluefin tuna クロマグロ
もどる