2. 様々なデータベースと配列解析
2-2.遺伝子領域、機能の予測 その2
■ NCBI ORF Finder を用いた遺伝子予測
【ORF:Open Reading Frame(オープンリーディングフレーム)】
まず、NCBI のページを開き、
左のメニューの"All Resources (A-Z)から "Open Reading Frame Finder" を探します。
塩基配列を入力し、"OrfFind" ボタンをクリックします。
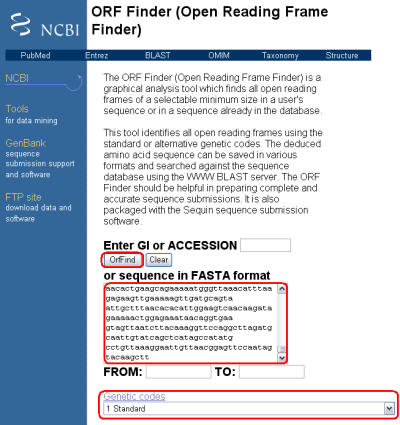
ミトコンドリアのように標準的な遺伝コードと異なる場合は、"Genetic code" のプルダウンメニューから
遺伝コードを指定する必要があります。
予測結果
塩基配列上のどの辺りに遺伝子が存在すると予測されたかが図として表示されています。 6本の帯は、ORF の読み枠が上から順に、+1、+2、+3、-1、-2、-3 となっています。読み枠 +1 は、 入力した塩基配列の1塩基目からアミノ酸へ翻訳を行う読み枠のことです。また読み枠 -1 は、 入力した塩基配列の最後の塩基から逆鎖の方向でアミノ酸に翻訳する読み枠です。
画面右側は、ORF の情報が長い順に表示されています。
赤枠で示したような水色の部分をクリックするとその領域のアミノ酸配列が表示されます。
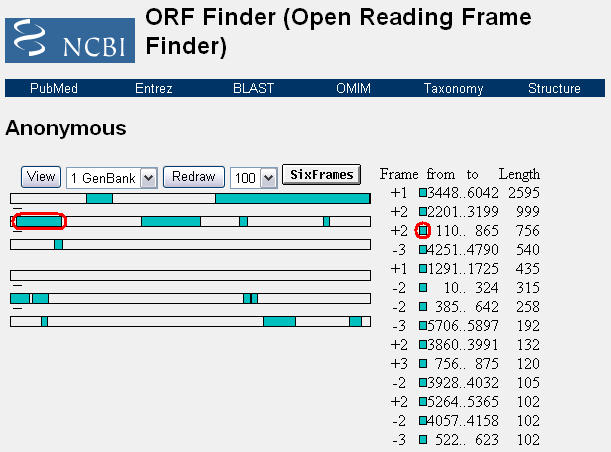
デフォルトの設定では、100塩基以上の ORF のみが表示されるようになっていますが、 赤枠のプルダウンから50または300などの数字を選択することができます。
また、表示されているアミノ酸配列をすぐに BLAST 検索にかけることもできます。

■ GenScan による真核生物での遺伝子予測
ACCESSION NG_012043 で登録されている GENE にコードされているアミノ酸配列を取得してみよう
(NCBI) 。
アミノ酸配列の予測
- reference sequence details をクリックし、配列情報へジャンプし、FASTA から配列を取得する。
- GENSCAN にアクセスし、配列を解析する。
アミノ酸配列の確認
- reference sequence details をクリックし、GenBank へジャンプし、CDS 情報から protein id を取得する。
- protein id からアミノ酸配列情報を取得する。
■ 転写産物との配列比較(塩基配列)
実際に発現されている mRNA の配列を決定することで、ゲノム配列上での遺伝子領域を調べます。
現在のシーケンス技術では一度のシーケンスで1kb程度の塩基配列しか決定することができない、
また、遺伝子の発現を、より網羅的に調査したいという研究背景があります。これらのことから、
cDNA の全配列を決定せずに、5' 末端と 3' 末端のみの配列だけをシーケンスした塩基配列を目印として
遺伝子が調べられています。このような配列は、EST (Expressed Sequence Tag)と呼ばれています。
現在では、非常に多くの EST 配列が決定されており、NCBI で蓄積されています。
■ 相同性検索(塩基配列、アミノ酸配列)
近縁の生物間では配列の相同性が高いという特徴があります。この特徴からゲノム配列の中で 配列相同性の高い領域を遺伝子として予測することができます。
相同性検索プログラムとしては、BLAST が最もよく利用されています。その他に感度の良いプログラムとして
PSI-BLAST や PHI-BLAST なども利用されています。
■ モチーフ検索(アミノ酸配列)
複数のタンパク質間において共通に見られる部分配列の特徴的なパターンのことをモチーフと呼びます。
モチーフのデータベースとしては、Pfam、PROSITE などが有名です。
-
Pfam (http://pfam.sanger.ac.uk/)
タンパク質配列ドメインと、配列上良く保存されている部分のマルチプルアライメントのデータベースです。 ドメインのマルチプルアライメントをもとに、プロファイルを作成し、このプロファイルを用いて、 配列データベースから類似配列をさらに検索しています。これらの配列がファミリーを形成しています。 -
PROSITE (http://us.expasy.org/prosite/)
UniProt から同一のファミリーに属する全タンパク質を抽出し、マルチプルアライメントを行うことで、 保存部位を特定しています。その保存部位から配列パターンを抽出し、その配列パターンを UniProt 全体に 適用したときに、できるだけ元のファミリーの配列だけが見出せるように配列パターンを洗練し、 その結果をデータベース化しました。
最近では、様々なモチーフデータベースサイトに一度の入力で検索を行えるシステムが公開されています。
代表的なサイトは、GenomeNet の Motif 検索サービスとヨーロッパの EBI の InterPro がよく利用されています。
本演習では、InterPro を利用してモチーフ検索を行います。
| システム名 | 開発 | 検索対象モチーフデータベース | 管理・運営 |
|---|---|---|---|
| Motif | GenomeNet | PROSITE、BLOCKS、ProDom、PRINTS、Pfam | motif.genome.jp/ |
| InterPro | EBI | PROSITE、ProDom、PRINTS、Pfam、SMART | www.ebi.ac.uk/interpro/ |
InterPro を利用してモチーフ検索を行います。
まず、GenomeNet にアクセスし、 DNA に結合する初期成長応答遺伝子(Early Growth Response Gene[EGR])のアミノ酸配列を入手します。 データベースとして UniProt を選択し、EGR 遺伝子の ID である egr1_human を入力、検索し、配列を入手しましょう。
続いて、どのようなモチーフが含まれているかを InterPro で調べましょう。
左メニューの "InterProScan" をクリックし、配列を貼り付けて、"Submit Job" ボタンをクリックします。
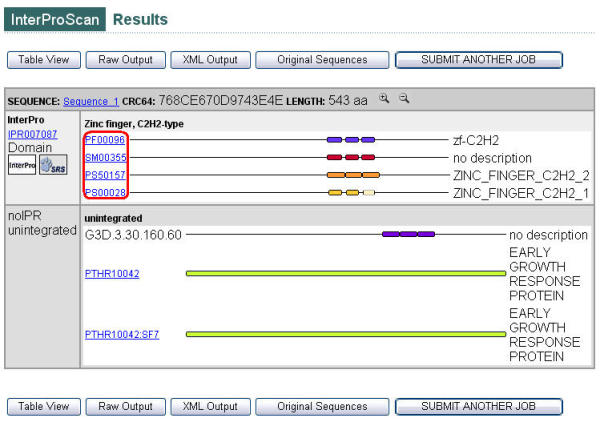
PDxxxxx → ProDom
PFxxxxx → Pfam
PRxxxxx → PRINTS
PSxxxxx → PROSITE
SMxxxxx → SMART